Hallan fósiles de cromosomas antiguos
Un equipo internacional de investigación con participación de la UAB ha descubierto fósiles de cromosomas antiguos en la piel de un mamut lanudo que murió hace 52.000 años. El hallazgo científico permite ensamblar genomas de especies extinguidas.
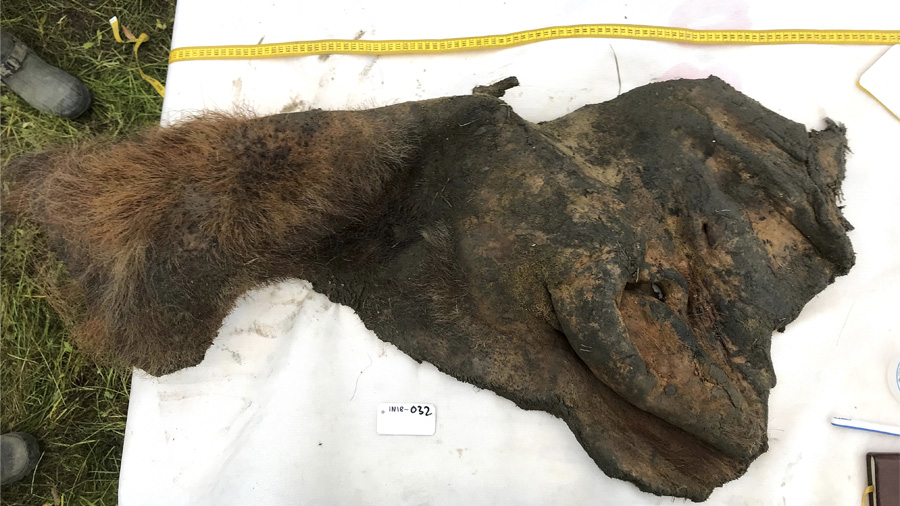
El estudio, publicado en la revista Cell, ha sido liderado por el Centro Nacional de Análisis Genómico (CNAG) y el Centro de Regulación Genómica (CRG), el Baylor College of Medicine (Estados Unidos) y la Universidad de Copenhague (Dinamarca). Por parte de la UAB ha participado Aurora Ruiz-Herrera, investigadora del Departamento de Biología Celular, Fisiología e Inmunología y del Instituto de Biotecnología y Biomedicina (IBB).
La investigación ha evidenciado que los pequeños fragmentos de ADN antiguo pueden sobrevivir durante un muy largo período de tiempo. Lo que han hallado los investigadores es, además, una muestra en la que la disposición tridimensional de estos fragmentos de ADN se congeló, por lo que permaneció intacta durante decenas de milenios.
Los cromosomas fósiles son una nueva herramienta muy potente para estudiar la vida en la Tierra. Esto se debe a que los fragmentos típicos de ADN antiguo rara vez superan la extensión de 100 pares de bases, o 100 letras de código genético, mucho más cortos que la secuencia completa de ADN de un organismo, que a menudo es de miles de millones de letras. En cambio, los cromosomas fósiles pueden abarcar cientos de millones de letras genéticas.
Los investigadores han determinado el número de cromosomas del mamut. «Encontramos que tienen 28 pares de cromosomas, lo que tiene mucho sentido, porque eso es lo que tienen los elefantes modernos, los parientes vivos más cercanos del mamut lanudo», detalla Juan Antonio Rodríguez, coautor principal del estudio e investigador del CNAG y de la Universidad de Copenhague.
El hallazgo ha cambiado las reglas del juego porque hace posible ensamblar la secuencia completa de ADN de especies que se extinguieron y obtener información que no teníamos hasta ahora. «Nos permite comparar moléculas de ADN antiguo con las secuencias de ADN de especies modernas. Para este estudio, hemos realizado la comparación entre los datos del genoma de mamut con los del genoma de elefante», explica Aurora Ruiz-Herrera.
Genes activos
El examen de los cromosomas fósiles permitió ver qué genes estaban activos. Para la mayoría de los genes, el estado de actividad coincidió con lo que el equipo de investigación vio en la piel de los elefantes modernos, pero no siempre: hay genes clave que regulan el desarrollo del folículo piloso con un patrón de actividad totalmente diferente al de los elefantes.
Otra característica relevante observada fue la supervivencia de los bucles de cromatina, estructuras mapeadas en humanos por el equipo de Baylor hace solo diez años.
«La supervivencia de los bucles en estos cromosomas antiguos es quizá la parte más impresionante», señala Marc A. Martí- Renom, coautor correspondiente del estudio y líder del grupo de Genómica Estructural en el CNAG, así como en el CRG. «Los bucles de ADN, que solo miden 50 nanómetros, son importantes porque acercan las secuencias de ADN activadoras a sus objetivos genéticos. Por tanto, estos fósiles no solo nos muestran qué genes estaban activos, sino también por qué».
El equipo de investigación observó que los cromosomas estaban en un estado similar al de las moléculas en el vidrio, lo que podría explicar cómo esos fragmentos de ADN de cromosomas antiguos habrían podido sobrevivir en el permafrost siberiano durante 52.000 años con su estructura tridimensional intacta. La hipótesis de que se habrían visto sometidos a un proceso de “transición vítrea” similar al que se crea con el procesado de carne seca o cecina se vio validada con varios experimentos realizados con carne seca de ternera: a nanoescala, los cromosomas de la carne siempre permanecieron intactos.
Artículo de referencia: Marcela Sandoval-Velasco et al. «Three-dimensional genome architecture persists in a 52,000-year-old woolly mammoth skin sample» Cell. https://doi.org/10.1016/j.cell.2024.06.002