Bioinformàtica de la Diversitat Genòmica
Línies de Recerca
El grup Bioinformàtica de la Diversitat Genòmica té com a objectiu explicar la naturalesa de la variació del genoma i la seva relació amb la variació fenotípica i la forma física. Una nova dimensió dels estudis de variació genètica la proporcionen els genomes complets que s'estan desxifrant cada cop més (per exemple, Projecte 1000 Genomes ), així com noves dades d'alt rendiment procedents d'altres capes òmiques , com les dades transcriptòmiques i epigenòmices de l'ENCyclopedia Of Dna. Elements (ENCODE/modENCODE) o l'International Human Epigenome Consortium (IHEC). Seguim un enfocament interdisciplinari, fusionant mètodes i coneixements de la genòmica, la genètica de poblacions, la ciència de dades, la biologia de sistemes i la bioinformàtica, per abordar, tant en Drosophila com en humans, els diferents objectius plantejats.
Actualment tenim les següents línies de recerca:
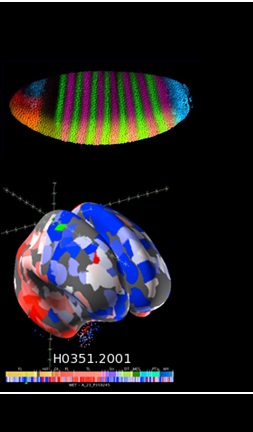
Mapeig de la selecció sobre desenvolupament d'embrions a Drosophila i cervell humà
Integrar informació de dades de genòmica de poblacions amb coneixements recents sobre genètica del desenvolupament ( Drosophila ), genòmica de cèl·lules unicel·lulars i transcriptòmica cerebral (humans) per obtenir proves de l'evolució del desenvolupament embrionari i l'evolució del cervell humà a nivell microevolutiu i provar la importància selectiva de la jerarquia en xarxes de gens, interaccions pleiotròpiques o evolució preferent de seqüències reguladores vs codificants.
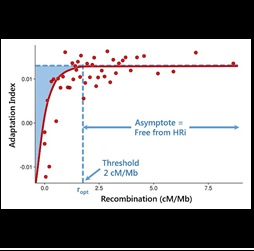
Definir paràmetres que mesuren el potencial adaptatiu d'un genoma
L'evidència recent basada en els patrons de diversitat del genoma mostra que molts loci i regions del genoma es veuen afectats per la selecció enllaçada recurrent (esborrany genètic). El nostre objectiu és desenvolupar un nou enfocament estadístic general per estimar la fracció de substitucions adaptatives de qualsevol regió del genoma, independentment de si estan experimentant o no esdeveniments selectius vinculats recurrents.

PopFly i PopHuman, els navegadors de genòmica poblacional de referència de Drosophila i humans
Actualitzeu els nostres navegadors amb noves dades genòmiques que s'han generat després de la publicació dels navegadors del genoma PopFly i PopHuman i complementeu-los amb dades d'altres espècies per convertir-los en els navegadors de genòmica poblacional de referència per als eucariotes.
Informació d'interès
Projectes finançats / Funded projects
Científics / Scientific
2018-2020: Genòmica poblacional de ladaptació. Investigadors: Mauro Santos (IP1) i Antonio Barbadilla (IP2), Alfredo Ruiz, Antoni Fontdevila, Pilar Garcia-Guerreiro, Francisco Rodríguez-Trelles, Sònia Casillas, Raquel Egea, Marta Coronat, Sergi Hervàs, Jesús Murga. MINECO CGL2017-89160-P. 2018-2020 (En execució).
2018-2020: Genòmica, Bioinformàtica i Biologia Evolutiva. Grup recerca consolidat de la Generalitat de Catalunya ( 2017 SGR 1379. Alfredo Ruiz (IP), Mauro Santos, Antonio Barbadilla, Mario Càceres, Isaac Salazar. 2018-2020 (En execució).
2014-2017: La interpretació de la variació genòmica des de la seqüència nucleotídica al fenotip a Drosophila i humans. Investigadors: Antonio Barbadilla (IP1), Mario Càceres (IP2), Isaac Salazar, Sònia Casillas, Marta Puig, Raquel Egea, Carla Giner, Marta Coronada, Sergi Hervàs, Isaac Noguera, Ion Lerga. MINECO BFU2013-42649-P. 2014-2017 (Finalitzat)
2014-2016: Genòmica, Bioinformàtica i Biologia Evolutiva. Grup de recerca consolidat de la Generalitat de Catalunya ( 2014 SGR 1346). Alfredo Ruiz (IP), Mauro Santos, Antonio Barbadilla, Mario Cáceres, Isaac Salazar. 2014-2016.
2010-2013: Anàlisi genòmica del polimorfisme i la divergència a Drosophila melanogaster . Investigadors: Antonio Barbadilla (IP), Sònia Casillas, Esther Betrán, David Castellà, Miquel Ràmia, Maite Barrón. MICINN BFU2009-09504/BMC. 2010-2013. (Finalitzat)
2008-2012: Panell de referència genètica de Drosophila: un recurs comunitari per a l'estudi de la variació genotípica i fenotípica. IPs: Trudy Mackay, Stephen Richards i Richard Gibbs. http://flybase.org/static_pages/news/whitepapers/Drosophila_Genetic Reference Panel_Whitepaper.pdf
2009–2013: Genòmica, bioinformàtica i evolució. Grup de recerca consolidat de la Generalitat de Catalunya ( 2009 SGR 88). Alfredo Ruiz (IP), Antonio Barbadilla, Roger Vila i Isaac Salazar. 2009-2013.
2007-2009: Patterns de nucleotide diversity in diferents functional regions de Drosophila and Chordates. Investigadors: Antonio Barbadilla (IP), Sònia Casillas, Natalia Petit, Raquel Egea, Esther Betrán. MEC BFU2006-08640/BMC. (Finalitzat)
2005-2008: Estudis associatius assistits per Inferència i Tecnologies Semàntiques (ASSIST). Coordinador del projecte: P. Mitkas (CERHT/ITI). Sisè Programa Marc. STRP: Informació biomèdica integrada per a una millor salut. FP6-IST-2004-027510. Soci IP UAB: Antonio Barbadilla (Finalitzat)
2005: Identificació de gens implicats en la susceptibilitat de patir fibromiàlgia i/o síndrome de fatiga crònica. Similituds i diferències genètiques. Fundació per a la Fibromiàlgia i la Síndrome de Fatiga Crònica, Fundació Echevarne, ebiointel, Dpt Genètica i Microbiologia UAB, CEGEN, banc ADN. (Finalitzat)
2003-2005: Genòmica comparada i funcional de Drosophila: causes i conseqüències de les reordenacions cromosòmiques naturals. Investigadors: A. Ruiz (IP), A. Barbadilla, Josefa González, Ferran Casal, Bàrbara Negre, Marta Puig, Cristina Santa, Sònia Casillas . MCYT - BMC2002-01708. 2003-2005. (Finalitzat)
1999-2002: Projecte: Distribució de les taxes de recombinació local causades per conversió i entrecreuament al llarg del genoma. Investigadors: A. Barbadilla (IP); A. Navarro i Cristina Santa. DGICYT - PB98-0900-C02-02. 1999-2002. (Finalitzat)
1996-1998: Origen i evolució molecular de les inversions cromosòmiques de Drosophila buzzatii. Investigadors: A. Ruiz (IP); A. Barbadilla; E. Betrán; A. Navarro; José María Ranz; Mario Càceres; Josefa González. DGICYT-PB95-0607. (Finalitzat)
1994-1996: Significat adaptatiu del polimorfisme cromosòmic. Investigadors: A. Ruiz (IP); A. Barbadilla; E. Betrán; A. Navarro. DGICYT – PB93-0844. (Finalitzat)
Tecnològics / Technological
2006-2012: Projecte Plataforma Bioinformàtica UAB-Instituts UAB-Instituts de Recerca Hospitalària / Plataforma Bioinformàtica UAB-Instituts de Recerca Hospitalària . A. Barbadilla (IP), S. Casillas, Àlex Sànchez, José Manuel Soria. Vicerectorat de Projectes Estratègics UAB, 2006-12. (Finalitzat).
2004-2005: Projecte Neotec/La Caixa per a l'execució del projecte ebioPlatform : / Neotec /La Caixa project for the developing of ebioPlatform . I. Durán, A. Barbadilla, B. Aranda, J. Puiggene, J. Colomer, N. Rierol. Ministeri dIndústria, 2004-05. (Finalitzat)
2002: Gestió del desenvolupament d'eines bioinformàtiques / Management for the development of bioinformatic tools . Investigador Principal (IP): A. Barbadilla. Conveni col·laboració Fundació Empresa i Ciència, UAB, 2002. (Finalitzat)
2001-2002: Projecte del Centre d'Innovació i Desenvolupament Empresarial (CIDEM) per a la creació d'una empresa de base tecnològica / CIDEM projecte per desenvolupar technological based company . Investigador Principal (IP): A. Barbadilla. 2001-02. (Finalitzat)
Difusió de la ciència / Communicating Science
Genètica i Genòmica: una invitació a la genètica. Projecte d'accions especials de difusió de la ciència i la tecnologia. Ministeri Ciència i Tecnologia. / Special action project for public communication of science. Investigadors: A. Barbadilla (Investigador Principal); C. Santa. AE00-0128-DIF. 2001-02. (Finalitzat)
Una invitación a la bioinformática. Any de la Computació. UAB 2008. IP: Antonio Barbadilla. (Finalizado).
Adapnation: melanogaster catch the fly . Sònia Casillas (IP), Robert Torres, Josefa González, Antonio Barbadilla. 2019-2020. Fundació espanyola per a la ciència i la tecnologia. FCT-19-14835.
Docents / Teaching
2019-2021 Adapta't . Projecte de millora de la qualitat docent subvencionat pel vicerectorat de Programació Acadèmica i de Qualitat de la UAB. Professors: Sònia Casillas (IP), Gemma Armengol, Antonio Barbadilla, Aina Colomer, Marta Coronat, Raquel Egea, Pilar Garcia, Ruth Gómez, Jesús Murga, Alicia Roque.
2011-2012: Coordinador Proposta i implantació Màster estratègic de bioinformàtica de la UAB . Acció estratègica del projecte UAB-Campus d'Excel·lència . (2011-2012)
2007-2009: Coordinador Proposta i implantació Grau Genètica de la UAB. Facultat de Biociències. (2007-2009)
2002-2019: Diseño e implementación una plataforma Web 2.0 para la docencia universitaria (actualización anual). Servei de Genòmica i Bioinformàtica (2002-2019)
2007-2008: Aula permanent de genètica. Ajuts projectes d'innovació docent 2007. Vicerectorat d'Estudis i de Qualitat, UAB. 2007-2008. Antonio Barbadilla (IP), Miquel Ràmia, Maite Barrón, Santiago Pastor, Ricard Marcos, Amadeu Creus, Antonia Velázquez. (Finalizado)
2004-2006: Implementació d'eines multimèdia en tota una Titulació: el cas de Biologia a la UAB / Implementació multimèdia tools a la Universitat de Col·legi: el cas de Biology a la UAB . (Conjuntament amb la comissió de docència de Biologia: Javier Retana, Amalia Molinero, Marina Luquín, Miquel Ninyerola i Elena Ibáñez). Projectes de millora de la qualitat docent AGAUR 2004MQD 00068. 2004-06. (Finalitzat)
Publicacions destacades
Rodriguez-Galindo, M., Casillas, S., Weghorn, D. and Barbadilla, A. 2020. Germline de novo mutation rates on exons versus introns in humans. Nat Commun 11, 3304 (2020). https://doi.org/10.1038/s41467-020-17162-z
Casillas*, S., R. Mulet*, P. Villegas-Mirón, S. Hervás, E. Sanz, D. Velasco, J. Bertranpetit, H. Laayouni & A. Barbadilla. 2018. PopHuman: the human population genomics browser. Nucl. Acids Res. gkx943, https://doi.org/10.1093/nar/gkx943
Salvador*, I., M. Coronado*, D. Castellano, A. Barbadilla & I. Salazar. 2018. Mapping selection within Drosophila melanogaster embryo’s anatomy. Molecular Biology and Evolution https://doi.org/10.1093/molbev/msx266.
Casillas, S. and A. Barbadilla. 2017. Molecular Population Genetics. Genetics 205: 1003–1035. https://doi.org/10.1534/genetics.116.196493
Huang, W. et al. 2014. Natural Variation in Genome Architecture. Among 205 Drosophila melanogaster Genetic Reference Panel Lines. Genome Research 2014. 24:1193-120.
T.F.C. Mackay*, S. Richards*, E.A. Stone*, A. Barbadilla*, ..., S. Casillas, M. Barrón, D. Castellano, P. Librado, M. Ràmia, J. Rozas et al. 2012. The Drosophila melanogaster Genetic Reference Panel: A Community Resource for Analysis of Population Genomics and Quantitative Traits. Nature 482: 173-178. * Equal contribution.
Egea, R., S. Casillas and A. Barbadilla (2008). Standard & Generalized McDonald and Kreitman test: a website to detect selection by comparing different classes of DNA sites. Nucleic Acids Res. 2008 July 1; 36 (Web Server issue): W157–W162.
Cáceres, M., J.M. Ranz, A. Barbadilla, M. Long & A. Ruiz. (1999). Generation of a widespread Drosophila inversion by a transposable element, Science 285: 415-418.
Totes les publicacions
Articles a International Science Journals (JCR)
Kapun, M. et al. 2021. Drosophila Evolution over Space and Time (DEST) - A New Population Genomics Resource. bioRxiv 2021.02.01.428994; doi: https://doi.org/10.1101/2021.02.01.428994
Rodriguez-Galindo, M., Casillas, S., Weghorn, D. and Barbadilla, A. 2020. Germline de novo mutation rates on exons versus introns in humans. Nat Commun 11, 3304 (2020). https://doi.org/10.1038/s41467-020-17162-z
Murga-Moreno, J.; Coronado-Zamora, M.; Hervás, S.; Casillas, S & A. Barbadilla 2019. iMKT: the integrative McDonald and Kreitman test. Nucleic Acids Research https://doi.org/10.1093/nar/gkz372
Coronado*, M, I. Salvador*, D. Castellano, A. Barbadilla & I. Salazar. 2019. Adaptation and conservation throughout the Drosophila melanogaster life-cycle. Genome, Biology and Evolution evz086, https://doi.org/10.1093/gbe/evz086.
Murga-Moreno, J.; Coronado-Zamora, M.; Bodelón, A.; Barbadilla, A. and Casillas, S 2019. PopHumanScan: the online catalog of human genome adaptation. Nucleic Acids Research, gky959, https://doi.org/10.1093/nar/gky959
Casillas*, S., R. Mulet*, P. Villegas-Mirón, S. Hervás, E. Sanz, D. Velasco, J. Bertranpetit, H. Laayouni & A. Barbadilla. 2018. PopHuman: the human population genomics browser. Nucl. Acids Res. gkx943, https://doi.org/10.1093/nar/gkx943 *Equal contribution.
Salvador*, I., M. Coronado*, D. Castellano, A. Barbadilla & I. Salazar. 2018. Mapping selection within Drosophila melanogaster embryo’s anatomy. Molecular Biology and Evolution https://doi.org/10.1093/molbev/msx266.
* Equal contribution.
S. Hervas, E. Sanz, S. Casillas, J. Pool and A. Barbadilla. 2017. PopFly: the Drosophila population genomics browser. Bioinformatics https://doi.org/10.1093/bioinformatics/btx301
Casillas, S. and A. Barbadilla. 2017. Molecular Population Genetics. Genetics 205: 1003–1035. https://doi.org/10.1534/genetics.116.196493
Pavlidou, P.; A. Daponte, R. Egea, E. Dardiotis, G.M. Hadjigeorgiou, A. Barbadilla & T. Agorastos. 2016. Genetic polymorphisms of FAS and EVER genes in a Greek population and their susceptibility to cervical cancer: a case control study. BMC Cancer 2016 16:923.
Castellano, D., M. Coronado-Zamora, JL. Campos, A. Barbadilla, A. Eyre-Walker. 2016. Adaptive evolution is substantially impeded by Hill-Robertson interference in Drosophila. Mol Biol Evol (2016) 33 (2): 442-455.
Guillén, Y. et al. 2015. Genomics of ecological adaptation in cactophilic Drosophila. Genome Biol Evol (2015) 7 (1):349-366.doi: 10.1093/gbe/evu291.
Huang, W. et al. 2014. Natural Variation in Genome Architecture. Among 205 Drosophila melanogaster Genetic Reference Panel Lines. Genome Research 2014. 24:1193-120.
A. Martínez-Fundichely, S. Casillas, R. Egea, M. Ràmia, A. Barbadilla, L. Pantano, M. Puig & M. Cáceres. 2014. InvFEST, a database integrating information of polymorphic inversions in the human genome. Nucl. Acids Res.42 (D1): D1027-D1032.doi: 10.1093/nar/gkt1122.
Gutiérrez F,M Gárriz,JM. Peri, L Ferraz,D Sol, JB Navarro, A Barbadilla, M Valdés. 2013. Fitness costs and benefits of personality disorder traits. Evolution and Human BehaviorFitness costs and benefits of personality disorder traits. Evolution and Human Behavior. Volume 34, Issue 1, January 2013, Pages 41–48.
T.F.C. Mackay*, S. Richards*, E.A. Stone*, A. Barbadilla*, ..., S. Casillas, M. Barrón, D. Castellano, P. Librado, M. Ràmia, J. Rozas et al. 2012. The Drosophila melanogaster Genetic Reference Panel: A Community Resource for Analysis of Population Genomics and Quantitative Traits. Nature 482: 173-178.
* Equal contribution.
Ràmia M, Librado P, Casillas S, Rozas, J & Barbadilla A. 2012. PopDrowser: The Population Drosophila Browser. Bioinformatics Feb 15;28(4):595-6.
Miquel M, López-Ribera I, Ràmia M, Casillas S, Barbadilla A, Vicient CM. 2011. MASISH: a database for gene expression in maize seeds. Bioinformatics 27(3):435-436
Petit, N. and A. Barbadilla (2009). The efficiency of purifying selection in Mammals vs Drosophila metabolic genes. J. Evol. Biology 22: 2118-2124.
Petit, N. and A. Barbadilla (2009). Selection efficiency and effective population size in Drosophila species. J. Evol. Biology 22: 515-526.
Egea, R., S. Casillas and A. Barbadilla (2008). Standard & Generalized McDonald and Kreitman test: a website to detect selection by comparing different classes of DNA sites. Nucleic Acids Res. 2008 July 1; 36 (Web Server issue): W157–W162.
Casillas, S., R. Egea, N. Petit, C. Bergman and A. Barbadilla (2007). Drosophila Polymorhism Database (DPDB): a portal for nucleotide polymorphism in Drosophila. Fly 1 (4): 205-211.
Casillas, S., A. Barbadilla and C. Bergman (2007). Purifying selection maintains highly conserved noncoding sequences in Drosophila. Mol Biol Evol doi:10.1093/molbev/msm150.
Petit, N., S Casillas, A. Ruiz and A. Barbadilla (2007). Protein polymorphism is negatively correlated with conservation of intronic sequences and complexity of expression patterns in Drosophila melanogaster. Journal of Molecular Evolution 64: 511-518.
Egea, R, S. Casillas, E. Fernández, MA Senar & A. Barbadilla (2007). MamPol: a database of nucleotide polymorphism in the Mammalia class. Nucleic Acids Res. 2007 January; 35(Database issue): D624–D629
Casillas, S, B Negre, A Barbadilla and A Ruiz (2006). Fast sequence evolution of Hox and Hox-derived genes in the genus Drosophila. BMC Evolutionary Biology 2006, 6:106.
Casillas, S & A. Barbadilla (2006). PDA v.2: improving the exploration and estimation of nucleotide polymorphism in large data sets of heterogeneous DNA. Nucl. Acids Res. 2006 34: W632-W634.
Casillas, S, N. Petit & A. Barbadilla (2005). DPDB: a database for the storage, representation and analysis of polymorphism in the Drosophila genus. Bioinformatics 2005 21: ii26-ii30; doi:10.1093/bioinformatics/bti1103
Bárbara Negre, Sònia Casillas, Magali Suzanne, Ernesto Sánchez-Herrero, Michael Akam, Michael Nefedov, Antonio Barbadilla, Pieter de Jong and Alfredo Ruiz (2005). Conservation of regulatory sequences and gene expression patterns in the disintegrating Drosophila Hox gene complex. Genome Research 15: 692-700, 2005
Casillas, S & A. Barbadilla (2004). PDA: a pipeline to explore and estimate polymorphism in large DNA databases. Nucleic Acid Research, Web Server issue 32: W166-W169.
Casillas, S., N Petit, R Egea, A Barbadilla. 2004. Storage, Representation and Analysis of Nucleotide Polymorphism. Spanish Bioinformatics Conference, 137-141.
Navarro, A. Barbadilla & A. Ruiz. (2000). Effect of inversion polymorphism on the neutral nucleotide variability of linked chromosomal regions. Genetics 155: 685-698.
Cáceres, M., J.M. Ranz, A. Barbadilla, M. Long & A. Ruiz. (1999). Generation of a widespread Drosophila inversion by a transposable element, Science 285: 415-418.
Cáceres, M., A. Barbadilla & A. Ruiz. (1999). Recombination rate predicts inversion size in Diptera», Genetics 153: 251-259.
Cáceres, M., A. Barbadilla & A. Ruiz. (1997). Inversion length and breakpoint distribution in the Drosophila buzzatii species complex: Is inversion length a selected trait?, Evolution 51: 1149-1155.
Betrán, E., J. Rozas, A. Navarro & A. Barbadilla. (1997). The estimation of the number and the distribution of gene conversion tracts from population DNA data», Genetics 146: 89-99.
Navarro, A., E. Betrán, A. Barbadilla & A. Ruiz. (1997). Recombination and gene flux caused by crossing over and gene conversion in inversion heterokaryotypes. Genetics 145: 281-295.
Ruiz A., J.M., Ranz, M. Caceres, C. Segarra, A. Navarro & A. Barbadilla. (1997). Chromosomal evolution and comparative gene mapping in the Drosophila repleta species group. Genetics and Mol. Biol. 20: 553-565.
Barbadilla A, King LM, Lewontin RC. What does electrophoretic variation tell us about protein variation? Mol Biol Evol. 1996 Feb;13(2):427-32
Ruiz, A. y A. Barbadilla. (1995). The contribution of quantitative trait loci and neutral marker loci to the genetic variances and covariances among quantitative traits in random mating populations», Genetics 139: 445-455.
Barbadilla, A. Ruiz, M. Santos & A. Fontdevila. (1994). Mating pattern and fitness component analysis associated with inversion polymorphism in a natural population of Drosophila buzzatii, Evolution 48: 767-780.
Naveira, H, & A. Barbadilla. (1992). The theoretical distribution of lengths of intact chromosome segments around a locus held heterozygous with backcrossing in a diploid species, Genetics 130: 205-209.
Barbadilla, A., H. Naveira, A. Ruiz & M. Santos. (1992). The estimation of genotypic probabilities in an adult population by the analysis of descendants, Genetical Research 59: 131/137.
Santos, M., A. Ruiz, J. E. Quezada Díaz, A. Barbadilla and A. Fontdevila. 1992. The evolutionary history of Drosophila buzzatii. XX. Positive phenotypic covariance between field adult fitness components and body size. Journal of Evolutionary Biology 5:403-422.
Ruiz, A., M. Santos, A. Barbadilla, J. E. Quezada-Díaz, E. Hasson & A. Fontdevila. (1991). Genetic variance for body size in a natural population of Drosophila buzzatii, Genetics 128: 739-750.
Barbadilla, A. 1991. State of art of the problematic of higher taxa. Ameghiniana 28:295-301.
Barbadilla, A., J. E. Quezada Díaz, A. Ruiz, M. Santos and A. Fontdevila. 1991. The evolutionary history of Drosophila buzzatii. XVII. Double mating and sperm predominance. Genetics Selection Evolution 23:133 140.
Santos, M., A. Ruiz, A. Barbadilla, J. E. Quezada Díaz, E. Hasson and A. Fontdevila. 1988. The evolutionary history of Drosophila buzzatii. XIV. Larger flies mate more often in nature. Heredity 61:255 262.
Barbadilla, A. & H. Naveira. (1988). «The estimation of parental genotypes by the analysis of a fixed number of their offspring.», Genetics 119: 465-472.
Articles en llibres
Berry, A. & A. Barbadilla. (2000). «Gene conversion is a major determinant of genetic diversity at the DNA level», en Evolutionary Genetics from Molecules to Morphology. R. S. Singh y C. B. Krimbras (eds.) Cambridge University Press, NY
A. Barbadilla (2009). "Darwinismo y Creacionismo". Cent cinquanta anys després de L’origen de les espècies de Darwin, (A. Navarro y C. Segarra eds.). Treballs de la Societat Catalana de Biologia, 60 (2009) 245-253. Versión castellana aquí
A. Barbadilla (2008). "La recerca sobre el genoma humà". Enciclopèdia Catalana.
A. Barbadilla. (2003). "La Comunicación Social de la Ciencia e Internet" en La Ciencia es Cultura, págs. 177-180. Coordinador: Manuel Toharia, Edita: Soc. Gestión Museo de las Ciencias Príncipe Felipe de Valencia.
A. Barbadilla. (2000). "La selección natural: me replico, luego existo", en Evolución y filogenia de Artrópodos, pág. 605-612. SEA Vol. 26
A. Barbadilla. (1990). "La estructura de la Teoría de la Selección Natural", en Temas actuales de Biología Evolutiva, en A. Ruiz y M. Santos (Coordinadores), págs. 163-191. Publicaciones UAB, Barcelona.
Assajos i articles de divulgació
Barbadilla, A.; S. Casillas & A. Ruiz. 2019. La teoría neutralista de la evolución molecular, medio siglo después. Investigación y Ciencia. Págs. 52-59. Febrero 2019.
Servidors web i bases de dades de diversitat genètica
-
Murga-Moreno, J.; Coronado-Zamora, M.; Hervás, S.; Casillas, S & A. Barbadilla 2019. iMKT: the integrative McDonald and Kreitman test. URL: https://imkt.uab.cat/
-
Murga-Moreno, J.; Coronado-Zamora, M.; Bodelón, A.; Barbadilla, A. and Casillas, S. 2018. PopHumanScan: the online catalog of human genome adaptation. URL: http://pophumanscan.uab.cat
-
Casillas, S., R. Mulet, P. Villegas-Mirón, S. Hervás, E. Sanz, D. Velasco, J. Bertranpetit, H. Laayouni & A. Barbadilla. 2018. PopHuman: the human population genomics browser. URL: http://pophuman.uab.cat
-
PopFly: the Drosophila population genomics browser. S. Hervas, E. Sanz, S. Casillas, J. Pool and A. Barbadilla. 2017. URL: http://popfly.uab.cat
-
PopDrowser: The Population Drosophila Browser. Ràmia M, Librado P, Casillas S, Rozas, J & Barbadilla A. 2012. URL: http://popdrowser.uab.cat
-
Standard & Generalized McDonald and Kreitman test: a website to detect selection by comparing different classes of DNA sites. Egea, R., S. Casillas and A. Barbadilla. 2008. URL: http://mkt.uab.cat
- PDA: pipeline para explorar y estimar el polimorfismo de grandes bases de DNA / PDA: a pipeline to explore and estimate polymorphism in large DNA databases - Casillas, S. & A. Barbadilla. 2004. PDA: Pipeline Diversity Analysis. Casillas, S. & A. Barbadilla. 2006. PDA v.2: improving the exploration and estimation of nucleotide polymorphism in large data sets of heterogeneous DNA. URL: http://pda.uab.es
-
MamPol: Mammalia Polymorphism Database. Drosophila Polymorphism Database. Plataforma de representación y análisis de la Diversidad Genética en Mamíferos / MamPol: Mammalia Polymorphism Database. Egea, R, S. Casillas, E. Fernández, MA Senar & A. Barbadilla (2006). URL: http://mampol.uab.es/
-
Plataforma para la representación y análisis de la Diversidad Genética en Drosophila. URL: http://dpdb.uab.es / DPDB: Drosophila Polymorphism Database. Casillas, S. & A. Barbadilla. 2005.
Elevator Pitch IBB-UAB 2019 de Marta Coronado
Finançament
Informació complementària





